Bei allgemeinen PubMed-Recherchen (wie z.B. nach „Latex Allergy“) erhält man eine Unmenge an Treffern, die ohne weitere Eingrenzung oft ein unverdauliches Mass erreichen (hier 3.000 Zitate). Am Besten grenzt man nun die Suche weiter sachlich ein: Latex Allergy AND Pregnancy (50 Treffer). Steht eine sachliche Eingrenzung nicht zur Verfügung, weil man z.B. alles zur Latexallergie finden möchte oder noch gar nicht weiß, unter welchen Aspekten man Latexallergie studieren möchte, kann man auf formale Eingrenzungen ausweichen (den so genannten PubMed-„Limits“). Diese begrenzen die Suche u.a. nach Altersgruppen, Artikelart (Clinical Trial, RCT, Review, Twin Study), Sprache oder Aktualität. Diese Limits sind jedoch oft unbefriedigend, weil dabei immer das Riskio besteht, relevante Arbeiten „wegzulimitieren“.
Bei einem meiner letzten Hausbesuche kam nun der interessante Vorschlag auf, eine PubMed-Recherche auf wichtige, impaktstarke Zeitschriften eingrenzen, z.B. auf alle Klasse 1 – Zeitschriften eines Fachgebietes. Dies ist in der Tat möglich, da PubMed a) die allermeisten Zeitschriften mit Impact Faktor indexiert (z.B. 97% in der Kardiologie) und b) eine Suche nach den Zeitschriftentiteln zuläßt. Im Folgenden finden Sie die genaue Vorgehensweise, wie man eine PubMed-Recherche auf bestimmte Zeitschriften (hier diejenigen mit hohem Impact Faktor) beschränkt:
1. Zeitschriftensuche
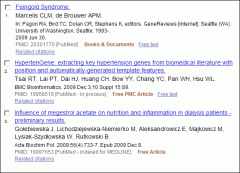
Man stellt sich zunächst in PubMed eine Suche zusammen, die alle Klasse 1 – Zeitschriften eines Fachgebietes umfasst. Die Zeitschriften werden am besten im NLM Catalog: Journals referenced in the NCBI Databases gefunden. Über den „PubMed search builder“ wird das Journal in die Suche übernommen. Nimmt man als Beispiel die Subject Category „Allergy“ (nur zugänglich an der Universität Münster), wären dies:
- Journal of allergy and clinical immunology (PubMed-Suche)
- Allergy (PubMed-Suche)
- Clinical and experimental allergy (PubMed-Suche)
- Contact Dermatitis (PubMed-Suche)
- Clinical Reviews in Allergy and Immunology (PubMed-Suche)
2. Aufsummierung
Im zweiten Schritt gilt es, die Artikel der einzelnen Journale durch eine OR-Verknüpfung aufzusummieren:
„J Allergy Clin Immunol“[Journal] OR „Allergy“[Journal] OR „Clin Exp Allergy“[Journal] OR „Contact Dermatitis“[Journal] OR „Clin Rev Allergy Immunol“[Journal]
3. Abspeichern
Diese Suche muss man nun dauerhaft abzuspeichern. Dies funktioniert entweder als Lesezeichen im Browser, dazu geht man rechts auf „Search Details“, klickt auf „see more“ und legt sich einen Lesezeichen (Bookmark) auf die angegebene URL.
Oder man benutzt seinen MyNCBI-Account (Registrierung kostenlos), klickt auf „Save Search“ (oben unter dem Suchfenster) und kann nun die Suche jederzeit durch Klick auf den gewählten Suchnamen in MyNCBI wieder aufrufen.
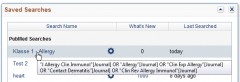
Anklicken zum Vergrößern
4. Eingrenzen
Will man irgendwann einmal seine Recherche (z.B. nach Latexallergien) auf diese Klasse 1- Zeitschriften eingrenzen, dann genügt es, die einmal abgespeicherte Suche über einen der beiden Wege aufzurufen und über „Advanced“ (oben unter dem Suchfenster) mit den aktuellen Suchtreffern (für Latexallergie) zu verknüpfen. Dies geschieht durch den Operator „AND“, siehe unten:

Anklicken zum Vergrößern
Will man sich die Mühe nicht machen (oder hat keine studentische Hilfskraft dafür an der Hand 😉 ), kann man sich – zumindest in der Inneren Medizin – mit folgendem Trick behelfen: Unter „Limits“ kann in PubMed das Trefferergebnis nach bestimmten „Subsets“ eingegrenzt werden, darunter AIDS, Cancer oder aber die 120 Journals des Abridged Index Medicus. Diese so genannten „Core Clinical Journals“ stellen die (aus angloamerikanischer Sicht) wichtigsten klinischen Zeitschriften dar, lassen aber eine eindeutige Qualifizierung nach Impact Faktoren vermissen.
Die Bibliothek hilft Ihnen aber gerne bei der Formulierung der Suchanfrage, einfach eine Email an info@zbmed.ms schreiben.