Forscher geben neue Einblicke in die Photosynthese
Die Photosynthese ist die wichtigste Grundlage des Lebens auf der Erde: Hierbei nutzen Pflanzen und einzellige Algen die Energie des Sonnenlichts und wandeln diese in Zucker und Biomasse um. Bei diesem Prozess wird Sauerstoff frei. Pflanzen-Biotechnologen der Universitäten Münster und Stockholm (Schweden) haben die Struktur eines neuen Proteinkomplexes aufgeklärt, der Energieumwandlungsprozesse in der Photosynthese katalysiert. Bei diesem Proteinkomplex handelt es sich um das Photosystem I, welches in Pflanzen als einzelständiger Proteinkomplex (Monomer) bekannt ist. Die Forscherinnen und Forscher um die Professoren Michael Hippler von der Westfälischen-Wilhelms-Universität (WWU) Münster und Alexey Amunts von der Universität Stockholm zeigten nun erstmals, dass zwei pflanzliche Photosystem-I-Monomere sich als Dimer zusammenlagern können und beschreiben die molekulare Struktur dieser neuartigen molekularen Maschine. Die Ergebnisse, die in der aktuellen Ausgabe der Fachzeitschrift „Nature Plants” veröffentlicht sind, ermöglichen molekulare Einblicke in den Photosynthese-Prozess in nie zuvor gesehener Präzision. Sie könnten helfen, die Reduktionskraft (also die Bereitschaft, Elektronen abzugeben) des Photosystems I beispielsweise zur Produktion von Wasserstoff als Energieträger in Zukunft effizienter zu nutzen.
Zum Hintergrund: Es gibt zwei Photosynthese-Komplexe, Photosysteme I und II genannt, die bei Licht unterschiedlicher Wellenlänge optimal arbeiten. Die Aufnahme von Lichtenergie in die Photosysteme I und II ermöglicht den Elektronentransport innerhalb der molekularen „photosynthetischen Maschine“ und treibt damit die Umwandlung von Lichtenergie in chemische Energie an. Dabei werden auch Elektronen vom Photosystem I auf das Protein Ferredoxin übertragen. In Grünalgen kann Ferredoxin bei der Photosynthese entstehende Elektronen auf ein Enzym namens Hydrogenase übertragen, welches dann molekularen Wasserstoff produziert. Dieser molekulare Wasserstoff entsteht also mithilfe von Lichtenergie, wird damit erneuerbar produziert und könnte als zukünftiger Energieträger dienen. Die Wissenschaftler fragten sich: Wie hängt die Produktion von photosynthetischem Wasserstoff mit der strukturellen Dynamik von monomerem und dimerem Photosystem I zusammen?

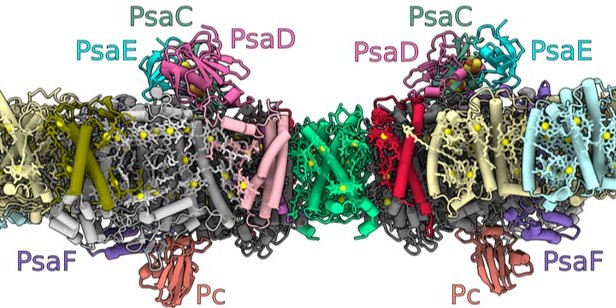
Das Photosystem-I-Homodimer aus der Grünalge Chlamydomonas reinhardtii besteht aus 40 Proteinuntereinheiten mit 118 Transmembran-Helices, die ein Gerüst für 568 Photosynthesepigmente bieten. Mithilfe von kryogener Elektronenmikroskopie zeigten die Forscher, dass die Abwesenheit der Untereinheiten mit den Bezeichnungen PsaH und Lhca2 zu einer Kopf-an-Kopf-Orientierung von monomerem Photosystem I (PSI) und seinen assoziierten Lichtsammel-Proteinen (LHCI) führt. Das Lichtsammel-Protein Lhca9 ist dabei das Schlüsselelement für die Vermittlung dieser Dimerisierung.
Die Wissenschaftler definieren in der Studie das genaueste verfügbare PSI-LHCI-Modell mit einer Auflösung von 2,3 Ångström (ein Ångström entspricht dem zehnmillionsten Teil eines Millimeters), einschließlich des flexibel gebundenen Elektronenüberträgers Plastocyanin, und weisen allen Pigmenten sowie 621 Wassermolekülen, die die Energieübertragungswege beeinflussen, die korrekte Identität und Orientierung zu.
Die gentechnisch vermittelte Herunterregulierung der Untereinheit Lhca2 führt im Zusammenhang mit dem Verlust eines zweiten Gens (pgr5) zu einer sehr effizienten Wasserstoffproduktion in der Doppelmutante. „Die Abreicherung von Lhca2 fördert die PSI-Dimer-Bildung, sodass wir daraus ableiten, dass die Hydrogenase bevorzugt photosynthetische Elektronen vom PSI-Dimer abgreift, wie in unseren früheren Arbeiten vorgeschlagen", betont Michael Hippler. „Die Struktur des PSI-Dimers ermöglicht uns nun durch gezielte gentechnische Eingriffe die Hypothese der verbesserten Wasserstoffproduktion durch das PSI-Dimer zu untersuchen.“
Originalveröffentlichung
Naschberger, A., Mosebach, L., Tobiasson, V., Kuhlgert, S., Scholz, M., Perez-Boerema, A., Ho, T.T., Vidal-Meireles, A., Takahashi, Y., Hippler, M., and Amunts, A. (2022). Algal photosystem I dimer and high resolution model of PSI:plastocyanin complex. Nature Plants; DOI: 10.1038/s41477-022-01253-4
Weitere Literatur
Ho et al. (2022), DOI: 10.1093/plphys/kiac055; siehe auch PCT Patent Application No. PCT/IL2021/051282 ″PHOTOSYNTHETIC MICROALGAE AND USE THEREOF FOR HYDROGEN PRODUCTION”
