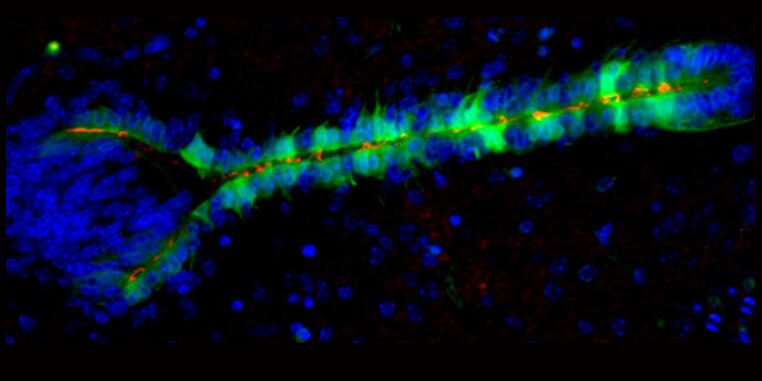
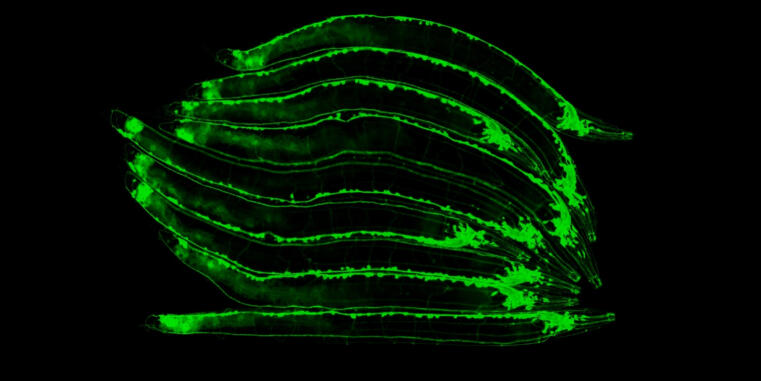
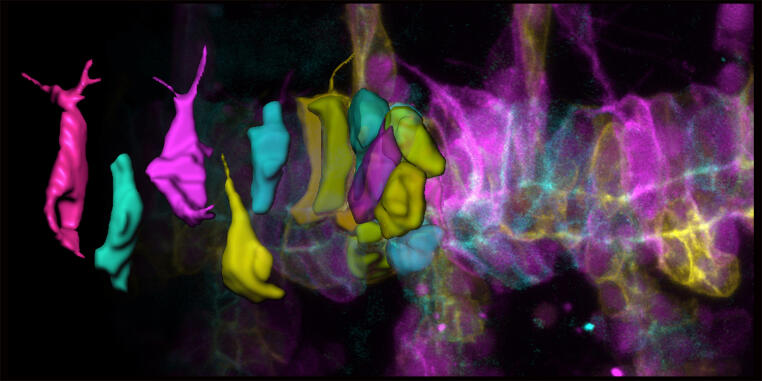
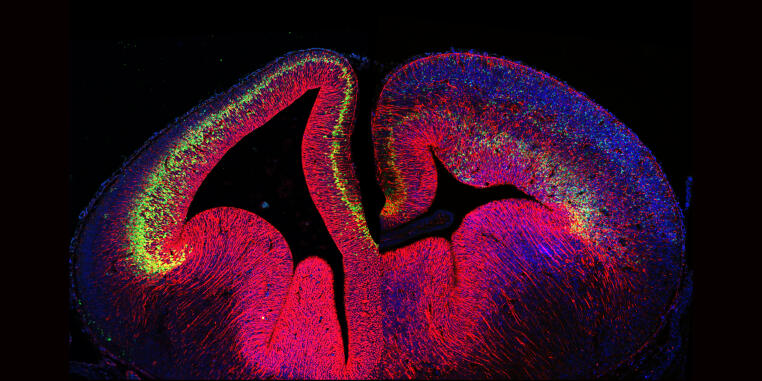






Das neu gegründete Institut für Integrative Zellbiologie und Physiologie (IIZP) ist aus dem Zusammenschluss des ehemaligen Instituts für Molekulare Zellbiologie (IMZ) und des Instituts für Zoophysiologie (IZP) hervorgegangen.
Dieses neue Institut entstand aus der Erkenntnis, dass physiologische und zellbiologische Prozesse eng miteinander verknüpft sind und nicht unabhängig voneinander verstanden werden können. Das IIZP hat die Mission, die komplexen Strukturen und physiologischen Prozesse in tierischen Zellen und Organismen auf unterschiedlichen Zeit- und Größenskalen zu untersuchen. Unser Ziel ist es, durch diesen Ansatz die molekularen, zellulären und biophysikalischen Prinzipien entwicklungsbiologisch, physiologisch und pathologisch relevanter Prozesse aufzuklären.
Die Forschungsarbeiten am IIZP integrieren Experimente an einzelnen Molekülen, isolierten Zellen, Geweben und lebenden Organismen. Dabei werden unterschiedliche Modellsysteme genutzt, die von Zellkulturen, Amöben, Krebstieren, Fadenwürmern und Fruchtfliegen bis hin zum Mausmodell reichen. Für unseren integrativen Forschungsansatz kombinieren wir biochemische und genetische Verfahren mit hoch-auflösenden, quantitativen Mikroskopie-Methoden.
Weitere Informationen finden Sie auf den Seiten unserer Forschungsgruppen.
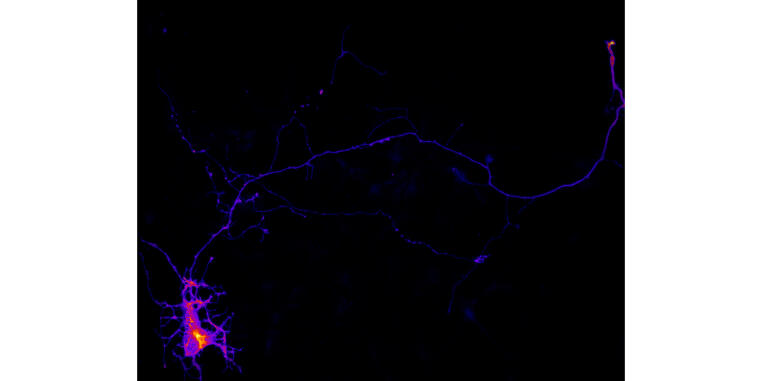
Mehr zur Arbeit finden Sie hier.
Forschungsförderung
Die Arbeit wurde durch die Deutsche Forschungsgemeinschaft (DFG) unterstützt.
Originalpublikation
Di Meo, D., Kundu, T. Ravindran, P., Shah, B, Püschel, A.W. (2024). Pip5k1γ regulates axon formation by limiting Rap1 activity. Life Sci Alliance 7: e202302383. doi: 10.26508/lsa.202302383.
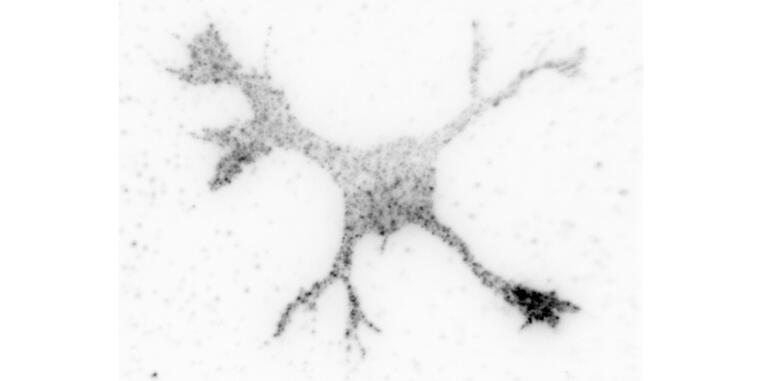
Mehr zur Arbeit finden Sie hier.
Forschungsförderung
Die Arbeit wurde durch die Deutsche Forschungsgemeinschaft (DFG) unterstützt.
Originalpublikation
Ravindran P, Püschel AW (2022). An isoform-specific function of Cdc42 in regulating mammalian Exo70 during axon formation. Life Sci Alliance: e202201722. doi: 10.26508/lsa.202201722.
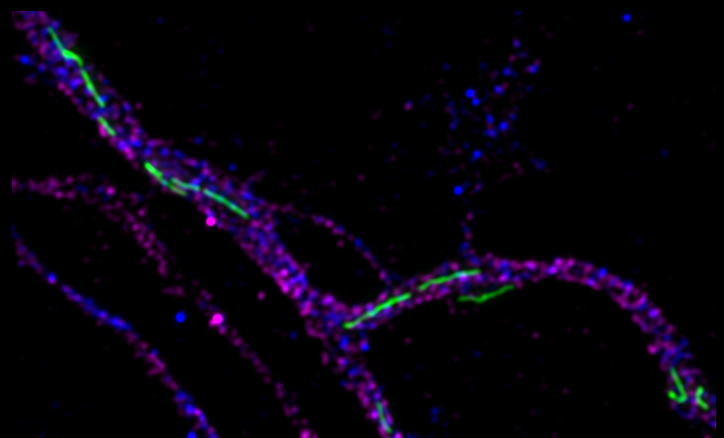
Mehr zur Arbeit finden Sie hier.
Forschungsförderung
Die Arbeit wurde durch die Deutsche Forschungsgemeinschaft (DFG) unterstützt.
Originalpublikation
Di Meo, D., Ravindran, P., Sadhanasatish, T., Dhumale, P., Püschel, A.W. (2021). The balance of mitochondrial fission and fusion in cortical axons depends on the kinases SadA and SadB. Cell Rep. 37, 110141. doi: 10.1016/j.celrep.2021.110141.
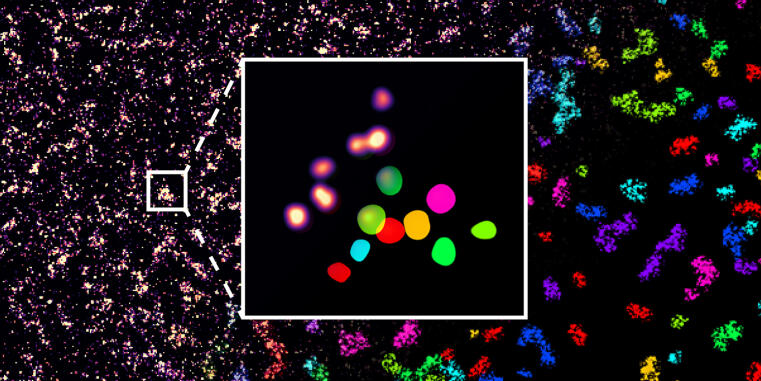
WWU Forscher optimieren Hochauflösungsmikroskopie-Anwendung zur Einzelmolekül-Detektion / Studie als Coverstory in ChemBioChem publiziert
Die Entwicklung der Hochauflösungsmikroskopie, die 2014 mit dem Nobelpreis ausgezeichnet wurde, erlaubt die Analyse zellbiologischer Prozesse mit einer Präzision von wenigen Nanometern, was sogar die Unterscheidung einzelner Moleküle innerhalb von Zellen möglich macht. Eine Limitation dieser Methode ist jedoch, dass in aller Regel die Zielmoleküle genetisch modifiziert werden müssen, um eine möglichst präzise Messung zu erreichen. Forschern an der WWU Münster haben nun ein Proteinmarkierungs-Verfahren optimiert, so dass auch native, nicht-modifizierte Proteine in ihrer natürlichen Umgebung auf der Einzelmolekülebene sichtbar gemacht und quantifiziert werden können.
Hintergrund und Methode
Hochauflösungsmikroskopie wird in der Regel mit einem Verfahren ermöglicht, bei dem durch eine kurzzeitige Anbindung von fluoreszent-markierten Proben Signale an einzelnen Molekülen erzeugt werden, wodurch deren genaue Lokalisation innerhalb der Zelle berechnet werden kann. Hierzu wird jedoch in aller Regel das zu untersuchende Molekül genetisch modifiziert. DNA-PAINT beruht zum Beispiel darauf, dass eine DNA-Bindestelle an das Zielprotein angebracht wird, an die wiederum komplementäre, fluoreszierende DNA-Stränge zur Detektion binden können. Andere Hochauflösungsmikroskopie-Methoden beruhen auf der Modifikation durch fluoreszente Proteine, die nach Wunsch an- und ausgeschaltet werden. Da solche genetischen Modifikationen in vielen Anwendungen aber unerwünscht und mitunter auch gar nicht möglich sind, bedarf es neuer Methoden zur Detektion nicht modifizierter, endogener Proteine.
Lisa Fischer, Doktorandin in der Arbeitsgruppe von Prof. Carsten Grashoff, entwickelte daher nun ein Verfahren, genannt Direct Peptide-PAINT, mit dem ein zentrales Zelladhäsionsprotein, Talin, mit einem fluoreszierenden Interaktions-Peptid markiert werden kann. Die erste Anwendung lieferte so nicht nur ersten Einblicke in die Verteilung dieses Moleküls in differenzierenden Stammzellen, es konnten auch erstmals einzelne Talin-Proteine in Gewebeschnitten sichtbar gemacht werden. Das neue Verfahren scheint daher geeignet, um die Untersuchung von Adhäsionsprozessen unter patho-physiologischen relevanten Bedingungen möglich zu machen. Die Forscher erwarten, dass dies wichtige, molekulare Einblicke in Krankheitsbilder erlaubt, denen eine dysfunktionale Zelladhäsion zu Grunde liegt.
Forschungsförderung
Die Arbeiten wurden durch die Deutsche Forschungsgemeinschaft (DFG) und dem Human Frontier Science Programm gefördert.
Originalpublikation
Lisa S. Fischer, Thomas Schlichthaerle, Anna Chrostek-Grashoff, and Carsten Grashoff. Peptide-PAINT Enables Investigation of Endogenous Talin with Molecular Scale Resolution in Cells and Tissues. Chembiochem. DOI: 10.1002/cbic.202100301