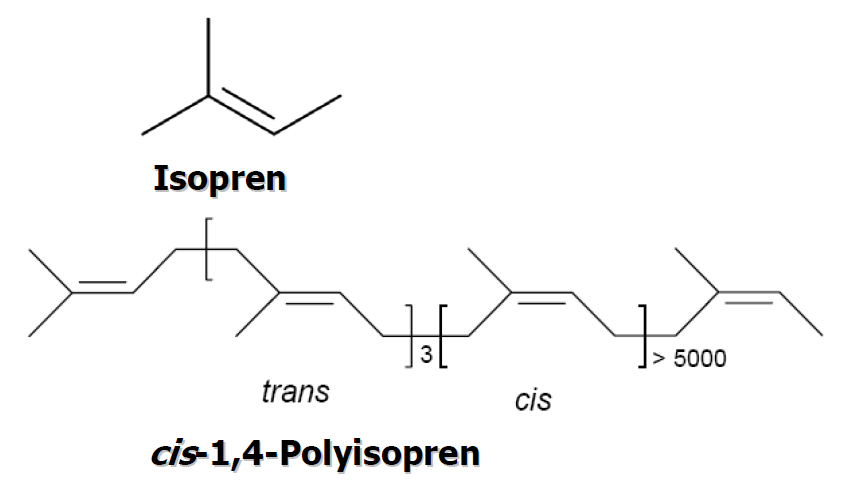
 Abb. 1 Strukturformeln der beiden Stereoisomere des Polyisoprens.
Abb. 1 Strukturformeln der beiden Stereoisomere des Polyisoprens.
Vorhaben und Ziele
Naturkautschuk wird aus dem Milchsaft (Latex) verschiedener tropischer Gewächse gewonnen, von denen Hevea brasiliensis die größte wirtschaftliche Bedeutung besitzt. Latex ist eine Emulsion von 0,5-1 µm großen Naturkautschuk-Tröpfchen. Weiterhin sind in geringen Gewichtsanteilen Proteine, Lipide und Kohlenhydrate enthalten, deren Gesamtanteil aber 6 % nicht übersteigt. Das Kautschuk-Polymer besteht aus cis-1,4-Polyisopren, einem hochmolekularen, ungesättigten Kohlenwasserstoff mit einem Polymerisationsgrad von 8.000 bis 30.000 Isopreneinheiten (Mw 0,5-2 x106). Um eine größere Elastizität und Zugfestigkeit der Kautschuk-Produkte zu erreichen, wird Kautschuk großindustriell dem 1840 von Goodyear entdeckten Prozess der Vulkanisation unterworfen; die Makromoleküle werden über Schwefelbrücken vernetzt.

Abb. 2 Kondomabbau durch Gordonia polyisoprenivorans
Erste systematische Untersuchungen zum mikrobiellen Abbau von Kautschuk wurden bereits 1914 von Söhngen und Fol durchgeführt, welche zur Isolierung einiger Actinomyceten führte, die Löcher im Kautschuk hervorriefen. Spence und van Niel (1936) isolierten Mikroorganismen, die auf Latex-Agarplatten eine Klärhofbildung zeigten. Erste Ergebnisse zum Mechanismus des Kautschukabbaus konnten durch Tsuchii (1985) aufgezeigt werden, der eine oxidative Spaltung der Doppelbindung unter Bildung von Carbonylenden vorschlägt.
Kautschuk-abbauende Mikroorganismen lassen sich in zwei Gruppen einteilen. Vertreter der klärhofbildenden Kautschukabbauer findet man ausschliesslich bei den Streptomyceten und verwandten Gattungen. Diese Gattungen sind der Öffentlichkeit vor allem durch die Produktion der wichtigen Stoffklasse der Antibiotika bekannt. So stammen 40 % aller bekannten Antibiotika von Vertretern der Gattung Streptomyces, wie z.B. Streptomycin, Tetracyclin, Erythromycin und Chloramphenicol.
Weniger bekannt, für das ökologische System aber immens wichtig, ist ihre Eigenschaft, verschiedene Polymere abzubauen. So besitzen viele Streptomyceten Enzyme zur Verwertung von Stärke, Chitin oder Cellulose. Daher war es für uns nicht verwunderlich, Kautschuk-verwertende Vertreter unter den Streptomyceten zu finden.

Abb. 3 Rasterelektronenmikroskopische Aufnahme von einem Latexhandschuh, der mit einem klärhofbildenden Streptomyceten inkubiert wurde
Da Streptomyceten genetisch nicht so leicht zugänglich sind wie etwa das "Haustierchen" der Mikrobiologen, Escherichia coli, war es erst einmal nötig, bestehende Transformationssysteme für die klärhofbildenden Streptomyceten zu optimieren bzw. neue zu entwickeln. Danach wurden auf chemischen Wege Mutanten erzeugt, die nicht mehr in der Lage waren, Latex zu verwerten. Diese Mutanten sollen dann mit Gesamt-DNA Fragmenten des Wildtyp-Stammes wieder zur Klärhofbildung befähigt werden.
Die zweite Gruppe von Bakterien zeigt adhäsives Wachstum auf kautschukhaltigen Materialien; sie benötigen den direkten Kontakt zum Kautschuk. Einige Vertreter dieser Gruppe wurden erstmals durch unsere Arbeitsgruppe isoliert und charakterisiert. Zu dieser Gruppe gehören ausschließlich nocardioforme Actinobacteria. Dieses sind Gram-positive Spezies mit einem hohen GC-Gehalt, welche Mycolsäuren synthetisieren. Viele Gattungen der Actinobacteria sind für ihre metabolische Vielfalt bekannt und können eine Vielzahl schwer zugänglicher Verbindungen zur Energiegewinnung nutzen. Stämme der Arten Gordonia polyisoprenivorans und Gordonia westfalica wurden als potente Kautschuk-Abbauer beschrieben und stehen als Vertreter der adhäsiv wachsenden Gruppe im Mittelpunkt der Forschungsarbeiten. Ein Ziel des Projekts ist die Aufklärung der biochemischen und molekularen Grundlagen des Abbaus von Polyisoprenoiden.
Abb. 4 Isolation von Mutanten auf Polyisopren-Sandwichplatten.
In unserer Arbeitsgruppe für diese Gattung wurde erstmalig ein Gen-Transfer-System etabliert und beschrieben. Aufbauend auf diesem Transfersystem wurden Untersuchungen zur Aufklärung der molekularen Mechanismen des mikrobiellen Kautschukabbaus durchgeführt. Dazu fand ein Transposon-basiertes Mutagenesesystem Anwendung, mit dessen Hilfe Mutanten, welche die Fähigkeit zum Kautschukabbau verloren haben, genetisch und wachstumsphysiologisch charakterisiert worden sind.

Abb. 5 Feststofffermenter unseres Kooperationspartners bioreact.
Auch deutet das Vorkommen von Megaplasmiden in Kautschuk-abbauenden Vertretern der Gattung Gordonia auf einen plasmidkodierten Kautschukabbau hin. Ein natives 101 kbp Plasmid, welches in Gordonia westfalica gefunden wurde, wurde zu diesem Zweck vollständig sequenziert. Da Mutanten, welche dieses Plasmid partiell verloren haben, nicht mehr in der Lage sind, Kautschuk zu verwerten, werden nun alle in Frage kommenden ORFs des Plasmids hinsichtlich ihrer Beteiligung am Kautschukabbau charakterisiert. Daneben konnte dieses Plasmid auch für die Konstruktion von geeigneten E. coli-Gordonia Shuttlevektoren genutzt werden.
Die Verfügbarkeit geeigneter Mikroorganismen und Kentnisse über den Polyisopren-Abbau ermöglichen neue biotechnologische Prozesse der Nutzung kautschukhaltiger Abfälle. In Zusammenarbeit mit der Deutschen Bundesstiftung Umwelt (DBU) wird ein mikrobiologisches Verfahren zur stofflichen Wiederverwertung kautschukhaltiger Rest- und Abfallstoffe entwickelt. Dabei werden auf der einen Seite Kautschukmaterialien wie Latexhandschuhe oder -matratzen in Feststofffermentern mikrobiologisch so umgesetzt, dass interessante Zwischenprodukte entstehen, die analysiert und für Produktionsprozesse eingesetzt werden können. Auf der anderen Seite wird an einer mikrobiologischen Entschwefelung von Altreifengranulaten gearbeitet, so dass ein größerer Anteil von diesem Material für die Produktion von Neureifen eingesetzt werden kann.
Stichwörter:
cis-1,4-Polyisopren, Latex, Naturkautschuk, Gummiabbau, Gordonia polyisoprenivorans, Gordonia westfalica, Micromonospora aurantiaca, Actinoplanes, Streptomyces, Streptomyces sp. K30, Megaplasmid
Publikationen
I Publications in peer reviewed journals (original contributions)
Arenskötter, M., D. Baumeister, M. Berekaa, G. Pötter, R.M. Kroppenstedt, A. Linos, and A. Steinbüchel. 2001. Taxonomic characterization of two rubber degrading bacteria belonging to the species Gordonia polyisoprenivorans and analysis of hyper variable regions of 16S rDNA sequences. FEMS Microbiol. Lett. 205:277-282.
Arenskötter, M., D. Baumeister, D. Bröker, U. Hölker, E. M. A. Ibrahim, J. Lenz, K. Rose, and A. Steinbüchel. 2003. Entwicklung eines biotechnologischen Verfahrens zur stofflichen Wiederverwertung kautschukhaltiger Rest- und Abfallstoffe. transkript Sonderband Biokatalyse pp. 28-32.
Arenskötter, M., D. Baumeister, R. Kalscheuer, and A. Steinbüchel. 2003. Identification and application of plasmids suitable for transfer of foreign DNA to members of the genus Gordonia. Appl. Environ. Microbiol. 69:4971-4974.
Arenskötter, M., D. Bröker, and A. Steinbüchel. 2004. Biology of the metabolically diverse genus Gordonia. Appl. Environ. Microbiol. 70:3195-3204.
Arenskötter, M., A. Linos, P. Schumann, R. Kroppenstedt, and A. Steinbüchel. 2005. Gordonia nitida Yoon et al. 2000 is a later synonym of Gordonia alkanivorans Kummer et al. 1999. Int. J. Syst. Evol. Microbiol. 55:695-697.
Arenskötter Q., J. Heller, D. Dietz, M. Arenskötter, and A. Steinbüchel. Cloning and characterization of {alpha}-methylacyl-coenzyme A racemase from Gordonia polyisoprenivorans VH2. Appl. Environ. Microbiol., 74(22):7085-7089, 2008.
Banh, Q., M. Arenskötter, and A. Steinbüchel. 2005. Establishment of Tn5096-based transposon mutagenesis in Gordonia polyisoprenivorans. Appl. Environ. Microbiol. 71:5077-5084.
Berekaa, M. M. and A. Steinbüchel. 2000. Microbial degradation of the multiple-branched alkane 2,6,10,15,19,23-hexamethyltetracosane (squalane) by Mycobacterium fortuitum and Mycobacterium ratisbonense. Appl. Environ. Microbiol. 66:4462-4467.
Berekaa, M.M., A. Linos, R. Reichelt, U. Keller, and A. Steinbüchel. 2000. Effect of pretreatment of rubber material on its biodegradability by various rubber degrading bacteria. FEMS Microbiol. Lett. 184:199-206.
Bröker, D., M. Arenskötter, A. Legatzki, D. H. Nies, and A. Steinbüchel. 2004. Characterization of the 101-kilobase-pair megaplasmid pKB1, isolated from the rubber-degrading bacterium Gordonia westfalica Kb1. J. Bacteriol. 186:212-225.
Bröker, D., D. Dietz, M. Arenskötter, and A. Steinbüchel. 2008. The genomes of the non-clearing-zone-forming and natural rubber-degrading species Gordonia polyisoprenivorans and Gordonia westfalica harbor genes expressing Lcp activity in Streptomyces strains. Appl. Environ. Microbiol. 74:2288-2297.
Ibrahim, E. M. A., M. Arenskötter, H. Luftmann, and A. Steinbüchel. 2006. Identification of poly(cis-1,4-isoprene) degradation intermediates during growth of moderately thermophilic actinomycetes on rubber and cloning of a functional lcp homologue from Nocardia farcinica strain E1. Appl. Environ. Microbiol. 72:2275-2282.
Rose, K., and A. Steinbüchel. 2002. Construction and intergeneric conjugative transfer of a pSG5-based cosmid vector from Escherichia coli to the polyisoprene rubber degrading strain Micromonospora aurantiaca W2b. FEMS Microbiol. Lett. 211:129-132.
Rose, K., K. B. Tenberge, and A. Steinbüchel. 2005. Identification and characterization of genes from Streptomyces sp. strain K30 responsible for clear zone formation on natural rubber latex and poly(cis-1,4-isoprene) rubber degradation. Biomacromolecules 6:180-188.
Rose, K., and A. Steinbüchel. 2005. Biodegradation of natural 1 rubber and related compounds: Recent insights into a hardly understood catabolic capability of microorganisms. Appl. Environ. Microbiol. 71:2803-2812.
Schulte C., M. Arenskötter, M. M. Berekaa, Q. Arenskötter, H. Priefert, and A. Steinbüchel. Putative involvement of an extracellular superoxidedismutase (SodA) in poly(cis-1,4-isoprene) degradation as radical scavenger? Appl. Environ. Microbiol., 74(24):7643-7653, 2008.
V.A.J. Kempf, M. Schmalzing, A.F. Yassin, K.P. Schaal, D. Baumeister, M. Arenskötter, A. Steinbüchel, and I. B. Autenrieth. 2004. Gordonia polyisoprenivorans septicemia in a bone marrow transplant patient. Eur. J. Clin. Microbiol. 23:226-228.
Linos, A., and A. Steinbüchel. 1998. Microbial degradation of natural and synthetic rubbers by novel bacteria belonging to the genus Gordonia. Kautschuk, Gummi Kunststoffe 51:496-499.
Linos, A., A. Steinbüchel, C. Spröer, and R. Kroppenstedt. 1999. Gordonia polyisoprenivorans sp. nov., a rubber degrading actinomycet isolated from automobile tire. Int. J. Syst. Bacteriol. 49:1785-1791.
Linos, A., M. M. Berekaa, U. Keller, R. Reichelt, J. Schmitt, H.-C. Flemming, R. M. Kroppenstedt, and A. Steinbüchel. 2000. Biodegradation of cis-1,4-polyisoprene rubbers by distinct actinomycetes: microbial strategies and detailed surface analysis. Appl. Environ. Microbiol. 66:1639-1645.
Linos, A., R. Reichelt, U. Keller, and A. Steinbüchel. 2000. A Gram-negative bacterium identified as Pseudomonas aeruginosa AL 98, is potent degrader of natural rubber and synthetic cis-1,4polyisoprene. FEMS Microbiol. Lett. 182:155-161.
Linos, A., M.M. Berekaa, A. Steinbüchel, K.K. Kim, C. Spröer, and R. Kroppenstedt. 2002. Gordonia westfalica sp. nov., a novel rubber degrading actinomycete. Int. J. Syst. Evol. Microbiol. 52:1133-1139.
Warneke, S., M. Arenskötter, K. B. Tenberge, and A. Steinbüchel. 2007. Bacterial degradation of poly(trans-1,4-isoprene) (gutta percha). Microbiology 153:347-356.
Yikmis, M., M. Arenskötter, K. Rose, N. Lange, H. Wernsmann, L. Wiefel, and A. Steinbüchel. 2008. Secretion and transcriptional regulation of the latex-clearing protein, Lcp, by the rubber-degrading bacterium Streptomyces sp. strain K30. Appl. Environ. Microbiol. 74:5373-5382.
II Other original contributions
Arenskötter, M., D. Baumeister, D. Bröker, E. M. A. Ibrahim, K. Rose, and A. Steinbüchel. 2002. Mikrobieller Abbau von Natur- und Synthesekautschuk. BIOforum 3/2002:124-126.
Linos, A., and A. Steinbüchel. 1996. Investigations on the microbial breakdown of natural and synthetic rubbers. In: DECHEMA Monographs, Vol. 133 (Eds. W. Sand und G. Kreysa), pp. 279-286.
III Review articles in peer reviewed journals or books
Arenskötter, M., D. Bröker, and A. Steinbüchel. 2004. Biology of the metabolically diverse genus Gordonia. Appl. Environ. Microbiol. 70:3195-3204.
Linos, A., and A. Steinbüchel. 2001. Biodegradation of natural and synthetic rubbers. In: Koyama and A. Steinbüchel (eds.) Biopolymers – Vol. 2, 1st Edition, Wiley-VCH, Weinheim, pp. 321-359.
IV Other review articles
Rehm, B. H. A., N. Hoffmann, Q. Qi, S. Fiedler, and A. Steinbüchel. 2004. Biosynthesis of latex-like polyhydroxyalkanoates. Proceedings of the "International Symposium on Bioconversion of Renewable Raw Materials" in Braunschweig, pp. 163-175.

